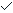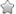最近开始接触单细胞数据,网上也有很多学习资料,琳琅满目,我也挑了一些视频资料进行学习,不过感觉还是需要进行实战训练才能更好地掌握这些知识,所以选了一篇2021年发表在nature communications的文章进行学习。
文献:
Single-cell RNA sequencing reveals functional heterogeneity of glioma-associated brain macrophages
GSE:GSE136001
一、数据下载并整理
下载数据:https://www.ncbi.nlm./geo/query/acc.cgi?acc=GSE136001


整理数据
- 每个样本都有barcodes, features和matrix三个文件,这是cellranger的标准三个文件。
- 每个样本单独新建一个文件,由于文章需要区分性别和组别,所以命名如下图。
- 每个样本里的三个文件分别重命名如下图。(Read10X函数读取必需这样命名才能读入)

上面的操作也可以通过下面的R代码来实现
fs = list.files('./GSE136001_RAW/',pattern = '^GSM')
samples <- substr(fs,1,10)
lapply( unique(samples), function(x){
y = fs[grepl(x,fs)]
folder = paste0('./GSE136001_RAW/',strsplit(y[1],split = '-f')[[1]][1])
#创建文件夹
dir.create(folder,recursive = T)
#重命名子文件夹并移动到相应的文件夹中
file.rename(paste0('./GSE136001_RAW/',y[1]),file.path(folder,"barcodes.tsv.gz"))
file.rename(paste0('./GSE136001_RAW/',y[2]),file.path(folder,"features.tsv.gz"))
file.rename(paste0('./GSE136001_RAW/',y[3]),file.path(folder,"matrix.mtx.gz"))
})
二、读入数据
加载R包
if(!require(BiocManager)) install.packages("BiocManager",update = F,ask = F)
if(!require(Seurat))install.packages("Seurat")
if(!require(Matrix))install.packages("Matrix")
if(!require(ggplot2))install.packages("ggplot2")
if(!require(cowplot))install.packages("cowplot")
if(!require(magrittr))install.packages("magrittr")
if(!require(dplyr))install.packages("dplyr")
if(!require(purrr))install.packages("purrr")
介绍两种方式读取原始数据(来自 cellranger 的基因/细胞计数矩阵)
一、Read10X函数批量读取
samples <- dir(path="./GSE136001_RAW/", pattern="^GSM")
for (i in samples){
assign(paste0("samples_raw_data", i), Read10X(data.dir = paste0("./GSE136001_RAW/", i)))
}
二、通过barcodes, features和matrix三个文件进行批量读取
samples <- dir(path="./GSE136001_RAW/", pattern="^GSM", full.names=T, recursive=T, include.dirs=T)
samples_raw_data <- lapply(samples, function(s) {
matrix_dir = s
# 设置barcodes, features和matrix三个文件的路径
barcode_path <- paste0(matrix_dir, "/", "barcodes.tsv.gz")
features_path <- paste0(matrix_dir, "/", "features.tsv.gz")
matrix_path <- paste0(matrix_dir, "/", "matrix.mtx.gz")
# 读取表达矩阵,基因名和barcodes
mat <- readMM(file = matrix_path)
feature_names = read.delim(features_path,
header = FALSE,
stringsAsFactors = FALSE)
barcode_names = read.delim(barcode_path,
header = FALSE,
stringsAsFactors = FALSE)
# 设置行名和列名
colnames(mat) = barcode_names$V1
rownames(mat) = feature_names$V2 # V1 - ENSMUSG, V2 - gene_names
# 输出gene/cell表达矩阵(行名为基因名,列名为细胞
mat
})
names(samples_raw_data) <- substr(samples, 17, nchar(samples))