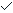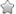近年来随着生物技术的不断发展,出现了许多克隆新基因的方法和手段,如图谱克隆技术、转座子标签技术、mRNA差异显示技术二基因组减法技术以及cDNA文库筛选技术等。但上述方法人多具有实验周期长、技术步骤烦琐且工作量大等特点。cDNA末端快速扩增技术(rapid amplification of cDNA ends, RACE)是一种基于PCR从低丰度的转录本中快速扩增cDNA的5'和3’末端的有效方法,以其简单、快速、廉价等优势而受到越来越多的重视。
经典的RACE技术是由Frohman等(1988)发明的一项技术,主要通过RT-PCR技术由已知部分cDNA序列来得到完整的cDNA5’和3’端,包括单边PCR和锚定PCR。该技术提出以来经过不断发展和完善,克服了早期技术步骤多、时间长、特异性差的缺点(Frohman等,1995:Schaefer,l995: Chen,1998: Bespalova等,1998: Matz等11999)。对传统RACE技术的改进主要是引物设计及RT-PCR技术的改进:改进之一是利用锁定引物((lock docking primer)合成第一链cDNA,即在oligo(dT)引物的3'端引入两个简并的核苷酸【5'-Oligo(dT)16_30MN-3', M=A/G/C;N=A/G/C/T】,使引物定位在poly(A)尾的起始点,从而消除了在合成第一条cDNA链时oligo(dT)与poly(A)尾的任何部位的结合所带来的影响;改进之二是在5‘端加尾时,采用poly(C),而不是poly(A);改进之三是采用RNase H一莫洛尼氏鼠白血.病毒(MMLV)反转录酶或选择嗜热DNA聚合酶可能在高温h (60 度 -70度 )有效地逆转录mRNA,从而消除了5‘端由于高CC含量导致的mRNA 二级结构对逆转录的影响;改进之四是采用热启动PCR (hot start PCR)技术和降落PCR(touch down PCR)提高PCR反应的特异性。
随着RACE技术日益完善,目前己有商业化RACE技术产品推出,如CLONTECH的MarathoTM技术和SMART TM RACE技术术。邢桂春等(2001)先后使用上述两种试剂盒进行RACE反应,结果发现使用Marathon TM所得到的片断总是比采用SMARTsup]TMRACE试剂盒到所得到的片断短。其原因在于Marathon TM技术反转录反应往往不能真正达到mRNA的5’末端。所以认为,进行RACE反应应当优选SMARTTM RACE试剂盒。以下就国内目前应用最广的SMART TM RACE试剂盒为例,简要概述RACE技术的原理和操作过程。 SMARTTM 3‘-RACE的原理是:利用mRNA的3‘末端的poly(A)尾巴作为一个引物结合位点,以连有SMART寡核营酸序列通用接头引物的Oligo(dT)30MN作为锁定引物反转录合成标准第一链cDNA。然后用一个基因特异引物GSP1(gene specific primer,GSP)作为上游引物,用一个含有部分接头序列的通用引物UPM(universal primer,UPM)作为下游引物,以cDNA第一链为模板,进行PCR循环,把目的基因3‘末端的。DNA片段扩增出来。见figure 1
screen.width-333)this.width=screen.width-333" width=455 height=466 title="Click to view full 3.jpg (455 X 466)" border=0 align=absmiddle>
SMARTTM 5‘-RACE的原理是先是利用mRNA的3‘末端的poly(A)尾巴作为一个引物结合位点,以Oligo(dT)30MN作为锁定引物在反转录酶MMLV作用下,反转录合成标准第一链cDNA。利用该反转录酶具有的末端转移酶活性,在反转录达到第一链的5‘末端时自动加上3一5个(dC)残基,退火后(dC)残基与含有SMART寡核苷酸序列Oliogo(dG)通用接头引物配对后,转换为以SMART序列为模板继续延伸而连上通用接头(figure 2 )。然后用一个含有部分接头序列的通用引物UPM(universal primer,UPM)作为上游引物,用一个基因特异引物2(GSP 2 genespecific primer,GSP)作为下游引物,以SMART第一链cDNA为模板,进行PCR循环,把目的基因5‘末端的cDNA片段扩增出来。最终,从2个有相互重叠序列的3' / 5‘-RACE产物中获得全长cDNA,或者通过分析RACE产物的3‘和5‘端序列,合成相应引物扩增出全长cDNA。
screen.width-333)this.width=screen.width-333" width=458 height=439 title="Click to view full 2.jpg (458 X 439)" border=0 align=absmiddle>
我们在实验中发现,要做好RACE反应实验不容易,实际操作中仍存在不少困难。因此,对RACE反应条件进行反复摸索是实验必须要做地。这是因为:第一,在5‘-RACE包含了有3个连续的酶反应步骤(反转录、同聚物加尾和PC R扩增),每一步都可能导致失败;第二,扩增。DNA末端的特异性完全依赖锚定引物及扩增DNA模板样品不均一性,因而特异性一般很低,常呈现不清晰的成片条带或截短的产物背景。因此,使用RACE技术扩增得到的特异末端片段,所获得的重组克隆最好能够全部测序,以排除RACE实验结果中扩增产物假阳性和假阴性,最终有可能获得新基因的全序列。我们相信,
随着RACE的不断改进和完善,优化条件下PCR扩增效率和忠实性的提高及PCR产物克隆技术的迅速发展,RACE必将在基因克隆及基因表达研究中发挥越来越大的作用。
下面简单介绍一下RACE的另一种代表方法——Self-Ligation法
步骤:
1. 反转录(RT)反应。
2. Hybrid RNA的分解。
3. 单链cDNA的自身连接。
4. PCR扩增5’未知区域。
5. 目的DNA片段的切胶回收。
6. DNA序列测定。
原理见figure 3
screen.width-333)this.width=screen.width-333" width=603 height=610 title="Click to view full 4.JPG (603 X 610)" border=0 align=absmiddle>
下面我把两种kit的说明书传到馆藏文献,望需要的战友去下载(注意7月4日后将删除)。
BD clontech SMART RACE PCRkit:
>
自顶一下,欢迎作RACE的战友参加讨论!
请问你有没有用过invitrigen公司的GeneRace,我下周就要做5’ RACE了。看过说明书了,但有点担心,因为作不出来的话就无法作重组了。
抱歉,我没有,我用的是BD clontech Co的,到时有问题我们可以讨论。
我没有看过invitrogen Co的,能否传一份protocol,email:yshu3507@126.com
感谢yshu3507战友的专题讲座!
希望对RACE有兴趣的朋友积极参加讨论!对有意义的回帖加分从优!
本专题将置顶1个月!
invitrogen公司5’RACE的简介:传统的RACE会得到多个PCR结果,因为其所使用的cDNA来源于断裂及全长mRNA。研究人员必须鉴定每一个 PCR 产物以获得带有全长5′末端序列的产物。GeneRacer™ 只针对全长的5′加帽mRNA,从而节省了花费在筛选大量PCR产物上的时间。示意图简介了GeneRacerTM 的工作原理。RNA样品经CIP(小牛肠碱性磷酸酶)和TAP(烟草酸焦磷酸酶)处理,保证GeneRacer™ 寡聚RNA仅同全长转录本连接。然后使用SuperScriptⅡ逆转录酶对这些转录本逆转录,得到的cDNA做为PCR的模板。GeneRacer™ 寡聚RNA序列做为GeneRacerTM 5′引物结合位点,这样,只有具有全长5′末端的cDNA才会被扩增。使用GeneRacer步骤及高保真度Platinum® Taq DNA聚合酶,就可以确保得到最高效的实验结果。
screen.width-333)this.width=screen.width-333" width=420 height=614 title="Click to view full clip_image001.jpg (420 X 614)" border=0 align=absmiddle>
还想请教一下你对race后dna扩增的看法(race直接产物量少,不易测序)。我认为巢式pcr法不好,因为就算用的是高保真tag酶,错配率还是很高的,会影响测序结果。我觉得连接到质粒再转化细菌,再在细菌内扩增后再测序比较好,因为细菌内dna聚合酶有校读功能。
你说的很好,我们做的方法是,先分开扩两端,测序后,从两端设计引物,扩全长,这样用高保真tag酶,就像我们平时扩已知序列那样轻松了。
我收集的一些资料:希望对做race的战友有用。
invitrogen公司5’RACE的方法为寡核苷酸帽法(Oligo caping method),该方法由日本东京大学铃木等开发。其特点为大规模、高通量、高精确度。
1. 传统的cDNA克隆化技术的缺点:理想的克隆cDNA应包含模板mRNA的5'帽的结构以及3'多聚A尾的全长序列。 然而,传统的方法克隆的cDNA,多数情况下其5'末端为部分缺失状态。究其原因有二:一是在以mRNA模板进行逆转录过程中,逆转录酶在从模板mRNA中逆转录酶脱落,只能得到部分拷贝的cDNA产物;二是在进行逆转录过程中,用了部分5'末端被降解的mRNA为模板合成cDNA,因此在构建cDNA文库中得到的多数是5'末端部分缺失状态的cDNA。换言之,在构建cDNA文库的过程中,应用逆转录酶以及应用极易受降解的mRNA是不可避免的,因此5'末端部分缺失就不足为奇了。
2.全長cDNA的克隆:为了构建含全長的cDNA 文库,就有必要从含有多数为非全长的cDNA文库中挑选出全長cDNA的一个过程。如果我们有办法识别全長cDNA的5'端和3'端,就有可能挑选出全长cDNA。 mRNA的3'端中有一个多聚 A尾结构可作为明确的识别标记。事实上、传统的cDNA文库的构建过程,也正是利用了 mRNA 3'端这一特征性的标签(tag),用多聚T引物合成cDNA的第一链而构建的。在mRNA的5'端存在一个帽结构,而自然状态下的这一结构不能象3'端的多聚 A尾一样,直接作为一个识别标记。由此可以设想,把这一结构用某种序列来替换,使其变成一个象多聚 A尾一样的特征性的序列标签(sequence tag),就有可能作为一个识别标记,这就是寡核苷酸帽法作为 5'端的识别标记的思路。
3. 全長cDNA文库的构建:为了克服传统的方法构建cDNA文库中所存在的缺点,铃木等开发了用特征性的序列标签替换mRNA5'端的帽的新方法—寡核苷酸帽法(Oligo caping method)。这一方法所获得的mRNA 5'端有RNA寡核苷,3'端有多聚 A尾特征性,因此其两端均有特征性的序列标签作为识别标记,我们就可能构建只含有全长cDNA的文库。
4.本技术的意义:确定启动子区域精确的确定基因的转录起始位点。基因的启动子区域中含有许多重要的调控基因转录的DNA序列(顺式元件),若要阐明基因在转录水平上的调控机制,克隆启动子序列是必不可缺的关键一环。到目前为止,已阐明启动子序列以及它们的调控机制的基因数量非常有限。事实上,标准的真核生物有关的启动子数据库Eukaryotic Promoter Database中登录的基因启动子也不过数百个而已。这其中一个重要原因之一是许多基因未能确切的确定转录起始位点。S1核酸酶作图法、引物延伸法、5'RACE法等传统的转录位点的确定方法,技術要求比较高,且有时由于种种原因,难以确定确切的转录起始位点。而利用铃木等开发的寡核苷酸帽法构建的cDNA文库中,随机的克隆全長的cDNA,适合于大规模、精确的获得基因转录起始位点的信息。
yshu3507 wrote:
你说的很好,我们做的方法是,先分开扩两端,测序后,从两端设计引物,扩全长,这样用高保真tag酶,就像我们平时扩已知序列那样轻松了。
请问yshu3507战友,“先分开扩两端,测序”,race产物的量一般很少,直接测序够吗?
分开扩两端,也要先亚克隆,一般不推荐直接用产物测序,因为在测序的时候,两端50bp左右的信号不会太强,也就是测不准确,这样会影响你下一步设计引物扩全长。
多谢。明天我准备做对照实验了,有问题再请教您!
可惜2周前我的硬盘坏了,上面有好多我收集来的有关RACE及RNAi的资料,现在由得再找了。
很高兴有了这么一个RACE专题,我以前好像提议了,但是没有人响应呵呵,不过现在好了,借机会问个问题:
1 我用的是clontech的race kit
2 一对基因特异引物可以扩出目的片断(测序验证过)
3 可是用这对GSP分别做3‘5’RACE时,做了好多的克隆都没有拿到任何结果(即使是大家公认的好作的3‘端)
4 我根据扩出的中间片段设计另外的GSP(两对),同样没有拿到任何结果,克隆做了好多
5 用第一对GSP的3’5‘RACE产物做模板,再用4中的GSP做nest-pcr仍然没有拿到要的东西
这种情况,大家认为应该如何往下做,该作些什么,谢谢
我们的做法是:如果RCR产物不是很特异或没有目的条带的话,建议用nest PCR。在做5‘时,对RNA要求比较高,最好用新鲜的组织提,随即做逆转录,扩增。
做RACE PCR条带单一的不多,尽可能的回收与目的片段大小相近的条带。克隆的时候,一般kit上建议挑8-10个克隆,多一个,多一份希望,因为RACE,尤其是5’太难了,我作了约6个月。
yshu3507 wrote:6个月!吓死我了。我准备做2个月,看来希望渺茫啊。
因为RACE,尤其是5’太难了,我作了约6个月。
^_^,如果您顺的话,一个礼拜足矣!
我的怎么做不出来?我做3'RACE的方法与上面说的Smart RACE一样,只是我的RNA是经poly A polymerase 加A后得到的mRNA.但是作不出来,有没有人做过类似的实验。请给我一点建议啊。郁闷中!!
能不能把你的具体步骤贴出来,让大家都来给你分析一下问题可能出在哪儿.
yshu3507 wrote:
^_^,如果您顺的话,一个礼拜足矣!
同意,5'-RACE确实比较难,但也有顺利的时候,我上周做5'-RACE也一次就成功了,我觉得关键还是RNA 的质量,如果RNA模板质量不好,后面做得再认真也是白费!
同时现在可以通过seegene公司的DNA Walking SpeedUp⑩ Kit 快速得到未知的序列,要比5'-RACE容易一些,Kit的简介如下:
Introduction:As a third commercial application of Seegene's proprietary ACP (Annealing Control Primer) Technology maximizing PCR specificity, the DNA Walking SpeedUp⑩ Kit is directed to methods of using our unique DNA Walking ACP (DW-ACP) primer which is designed to capture unknown target sites and the optimized PCR conditions (referred to as DW ACP-PCR⑩ Technology hereunder). Due to the unique features of the DW-ACP primer system, DW ACP-PCR⑩ Technology enables the researchers to obtain only genuine unknown target products up to the length (up to 2 Kb) of which Taq polymerase is able to synthesize. This method provides the most powerful and revolutionary way to directly amplify unknown sequences adjacent to known sequences (Fig. 1). Whole genomic DNA, total RNA, cDNA or plasmid (clone) can be used as starting material.
The kit provides four different DW-ACP 1, 2, 3, 4 each having "GGTC" sequence at 3'
end which is present in every 1-2 kb template on average. To extend this, unknown
target sequences can be amplified using DW-ACPs and TSPs (Target Specific Primers).
具体的原理和方法见以下的网页:
>
看了一下,发现这种方法并不一定好。
1.Gene-Specific Linear Amplification of Second Strand cDNA,这一步为单引物PCR,随后用5‘端带一部分已知序列的N10随机引物进行延伸,因为是随机引物,故究竟能够延伸多长的未知区域就无从知道,后面即使采用多轮巢式PCR,我看也不一定能够得到单一条带,所以后面的工作量相当大,而且不一定有可靠的结果,万一提总RNA时碰上基因组污染就更麻烦了!
2.形成套索结构的难度较大,此步类似TaKaRa 5’ RACE Kit中利用5‘-P化引物进行自身环化,感觉还没有TaKaRa 5’ RACE Kit简洁,操作相当烦琐!
See-gene公司虽然已经推出了基于此法的替代RACE试剂盒,但我觉得该技术并不一定能够普及适用,为广大用户所接受!
希望有DX指正!
谢谢 immuno05 战友的高见,但老兄可能没有仔细看,seegene公司的DNA Walking SpeedUp⑩ Kit 和Universal Fast Walking (UFW) 还是有一定的区别,seegene公司的DNA Walking SpeedUp⑩ Kit 可以用DNA或者cDNA作为模板扩增基因的序列,据我所知它目前已经广泛被广泛使用,所以immuno05 兄的担心可能是多余的!
我用的是这个公司的试剂盒用了好几次都没有出现预期的条带,总是非特异的带,不是大了就是小了,我想向用过这个试剂盒的高手请教,想知道操作中及使用中的注意事项。特别是哪个试剂盒提供的反转录温度为42~55度,我想知道我究竟用哪个温度反转比较好呢,都做了很久了一直都没有结果,很是着急啊,眼看要毕业了!恳请各位大虾知道!
qq 147610423
hanxgan@tom.com
好贴,已收藏。:)
hanxgan wrote:
我用的是这个公司的试剂盒用了好几次都没有出现预期的条带,总是非特异的带,不是大了就是小了,我想向用过这个试剂盒的高手请教,想知道操作中及使用中的注意事项。特别是哪个试剂盒提供的反转录温度为42~55度,我想知道我究竟用哪个温度反转比较好呢,都做了很久了一直都没有结果,很是着急啊,眼看要毕业了!恳请各位大虾知道!
qq 147610423
hanxgan@tom.com
从mRNA扩增其UTR区,RT时的温度主要取决于你的Target Specific Primer的Tm,通常比预测的Tm低3-5度,当然,还要考虑逆转录酶的最适温度,譬如普通MMLV逆转录在不加一些岩藻糖等一些高温稳定剂时就不能用45度甚至更高的温度。
seegene公司的DNA Walking SpeedUp⑩ Kit 是根据一个韩国人的技术而开发成的,由于其原理上的固有缺陷,并不能完整扩增一个cDNA的5‘或3’端的未知区域。它先用一种类似于随机引物的Annealing control primer/DW-ACP的3‘端的序列来随机引发,进而通过几轮nested-PCR来扩增出一部分未知区域,请结合下面的原理图,这种方法是不可能P出全部的未知区域的!
注意:该技术的独特之处就在于它的DW-ACP引物,其5’为一段设计独特的专用于后面nested-PCR的序列,3‘端的几个碱基序列类似于一种随机序列,通过连接5‘端与3’端之间的一段poly( I )5来调节3‘端序列的随机引发位点,我认为,将该方法用于RACE,是不可能得到全部长度的的UTR区的。
就5’-RACE而言,我认为目前最理想还是SMART-RACE试剂盒,当然,价钱也是最贵的,就费效比而言,我觉得TaKaRa的基于IPCR的5‘-RACE试剂盒还是不错的!
(缩略图,点击图片链接看原图)
那位前辈有结合3’RACE 与5’RACE原理构建全长cDNA 文库的经验,或有相关资料请回复帮帮后生,在此感激不尽。
我们准备构建一种动物组织的全长文库,实验设计思路如下:
组织——提取总RNA——反转录合成cDNA 第一条链(反转录引物含有接头引物,有酶切位点)——RNase A & RNase H消化杂合cDNA 中RNA——TdTase加尾(dCTP 加tail c)——高保真Tap酶PCR扩增得到全长双链cDNA (P1:针对反转录引物中的接头引物设计;P2:针对加dtail 设计,含接头引物及8个G)——PCR产物与T-vector连接——转化得文库
我们实验中遇到的难点如下:
1高保真Tap酶PCR扩增得到全长双链cDNA ,PCR产物电泳理想情况下图谱应有什么特点?
2这种PCR产物我们电泳得到约200bp~30kb的拖尾条带,但经PCR产物纯化试剂盒纯化,再用乙醇沉淀,用双蒸水溶解沉淀以浓缩,电泳鉴定这种浓缩PCR产物时发现有大量(约90%)的DNA跑不出加样孔,该如何解释?
3这种PCR产物与T-vector连接,连接效率极低,都有那些因素会照成,怎样去避免?
恳请高手明鉴,或帮助收集相关资料给予回复!
谢谢各位大虾。我正打算做呢。
本人又将第二次PCR的产物跑了一次电泳,还是跑不出孔,不知道是怎么回事
我用的是TaKaRa的RACE试剂盒,
望各位顶力相住,
急!!!!!!!!!!!!!!!!!!!!